R语言与机器学习中的回归方法学习笔记
机器学习中的一些方法如决策树,随机森林,SVM,神经网络由于对数据没有分布的假定等普通线性回归模型的一些约束,预测效果也比较不错,交叉验证结果也能被接受。下面以R中lars包包含数据集diabetes为例说明机器学习中的回归方法。一、数据集及交叉验证办法描述 Diabetes数据集包含在R的lars包中,数据分为x,y,x2三个部分,因变量为y,数据是关于糖尿病的
机器学习中的一些方法如决策树,随机森林,SVM,神经网络由于对数据没有分布的假定等普通线性回归模型的一些约束,预测效果也比较不错,交叉验证结果也能被接受。下面以R中lars包包含数据集diabetes为例说明机器学习中的回归方法。
一、数据集及交叉验证办法描述
- n<-length(dataset)
- index1<-1:n
- index2<-rep(1:5,ceiling(n/5))[1:n]
- index2<-sample(index2,n)
二、回归树
1.选择一个属性放置在根节点,为每个可能的属性值产生一个分支
2.将样本划分成多个子集,一个子集对应于一个分支
3.在每个分支上递归地重复这个过程,仅使用真正到达这个分支的样本
4.如果在一个节点上的所有样本拥有相同的类别,即停止该部分树的扩展
1.ID3:Information Gain
2.C4.5:Gain Ratio
3.CART:Gini Index
rpart(formula, data, weights, subset,na.action = na.rpart, method,
主要参数说明:
fomula回归方程形式:例如
data数据:包含前面方程中变量的数据框(dataframe)。
na.action缺失数据的处理办法:默认办法是删除因变量缺失的观测而保留自变量缺失的观测。
method根据树末端的数据类型选择相应变量分割方法,本参数有四种取值:连续型“anova”;离散型“class”;计数型(泊松过程)“poisson”;生存分析型“exp”。程序会根据因变量的类型自动选择方法,但一般情况下最好还是指明本参数,以便让程序清楚做哪一种树模型。
parms用来设置三个参数:先验概率、损失矩阵、分类纯度的度量方法。
control控制每个节点上的最小样本量、交叉验证的次数、复杂性参量:即cp:complexitypamemeter,这个参数意味着对每一步拆分,模型的拟合优度必须提高的程度,等等。
prune(tree, cp, ...)
主要参数说明:
tree一个回归树对象,常是rpart()的结果对象。
cp复杂性参量,指定剪枝采用的阈值。cp全称为complexity parameter,指某个点的复杂度,对每一步拆分,模型的拟合优度必须提高的程度,用来节省剪枝浪费的不必要的时间,R内部是怎么计算的我不知道,希望读者能够补充。
运行代码:
- library(rpart.plot)
- reg<-rpart(y~.,w)
- rpart.plot(reg,type=2,faclen=T)
- w<-diabetes[,2:3]
- n<-length(w$y)
- index1<-1:n
- index2<-rep(1:5,ceiling(n/5))[1:n]
- index2<-sample(index2,n)
- NMSE<-rep(0,5)
- NMSE0<-NMSE
- for(i
in 1:5){ - m<-index1[index2==i]
- reg<-rpart(y~.,w[-m,])
- y0<-predict(reg,w[-m,])
- y1<-predict(reg,w[m,])
- NMSE0[i]<-mean((w$y[-m]-y0)^2)/mean((w$y[-m]-mean(w$y[-m]))^2)
- NMSE[i]<-mean((w$y[m]-y1)^2)/mean((w$y[m]-mean(w$y[m]))^2)
- }
> NMSE
[1] 0.7892592 0.8857756 0.8619379 1.0072968 0.7238316
> NMSE0
[1] 0.3503767 0.3417909 0.3400387 0.3192845 0.3467186
> reg$cptable
1
2
3
4
5
6
7
8
9
10 0.01089168
11 0.01070564
12 0.01042308
13 0.01003363
14 0.01000000
- reg2<-prune(reg,cp=0.025)
- rpart.plot(reg2,type=2,faclen=T)

> NMSE
[1] 0.5982049 0.6995054 0.6826815 0.8970573 0.6407927
> NMSE0
[1] 0.5559462 0.5177565 0.4953384 0.5019682 0.5233709
三、boosting回归
mboost(formula, data = list(),
baselearner = c("bbs", "bols", "btree", "bss", "bns"), ...)
其中formular需要使用到函数btree():
btree(..., tree_controls = ctree_control(stump = TRUE,
mincriterion = 0,
savesplitstats = FALSE))
试运行下列代码:
- library(mboost)
- reg<-mboost(y~btree(x2.ltg)+btree(x2.bmi)+btree(x2.bmi.ltg),
- data=w,control=boost_control(nu=0.1))
我们可以得到boosting回归的信息,通过plot函数还有可视化结果。

这里值得一提的是我在做boosting回归时为了减少工作量直接做了变量选择(这个选择与回归树的最终选择是一致的,所以也有一定的道理)。最后交叉验证的结果为:测试集NMSE为0.5513152,训练集NMSE为0.4656569。比起上次计算的回归树模型测试集NMSE为0.7036484,训练集NMSE为0.518876好了不少。
四、bagging回归与boosting回归想法类似,bagging回归的做法就是不断放回地对训练样本进行再抽样,对每个自助样本都建立一棵回归树,对于每一个观测,每棵树给一个预测,最后将其平均。 对diabetes数据做bagging回归,使用到的函数包为ipred,使用函数为bagging(),用法如下:
bagging(formula, data, subset, na.action=na.rpart, ...)
主要参数介绍: Formula:回归方程形式 Data:数据集 Control:对树枝的控制,使用函数rpart.control(),可以控制诸如cp值,xval等参量。 输入代码:
- library(ipred)
- reg<-bagging(y~.,data=w,coob=TRUE,control=rpart.control(cp=0.025))
结果为: Baggingregression trees with 25 bootstrap replications Call:bagging.data.frame(formula = y ~ ., data = w, coob = TRUE, control =rpart.control(cp = 0.025)) Out-of-bagestimate of root mean squared error:58.3648 使用交叉验证(代码略),得到结果:测试集NMSE为0.5705753,训练集NMSE为0.3906232。比起上次计算的回归树模型测试集NMSE为0.7036484,训练集NMSE为0.518876好了不少。 五、随机森林回归 与bagging回归相比,随机森林则更进一步,不仅对样本进行抽样,还对变量进行抽样。 对diabetes数据做随机森林回归,使用到的函数包为randomForest,使用函数为randomForest(),用法如下:
randomForest(formula, data=NULL, ..., subset, na.action=na.fail)
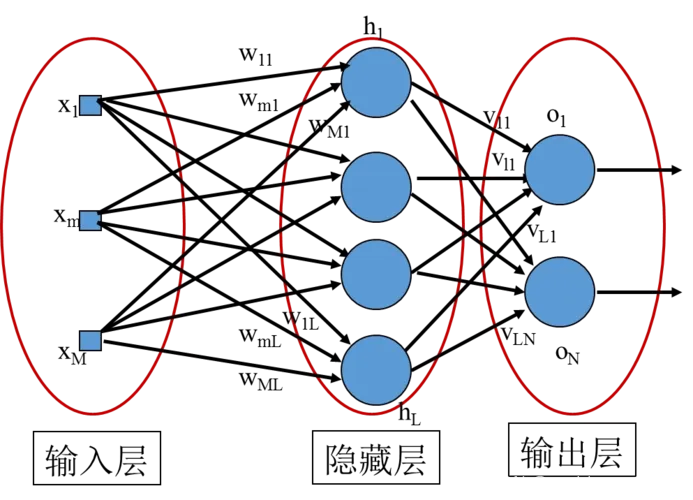
这里值得一提的是,随机森林有个十分牛逼的性质,不会出现过拟合现象,这也省去了我们确定树的分类程度等一系列麻烦的事情。得到结果:测试集NMSE为0.08992529,训练集NMSE为0.08835731,效果显著提升。随机森林还可以输出自变量重要性度量,试运行代码:
- library(randomForest)
- reg<-randomForest(y~.,data=w,importance=TRUE)
- reg$importance
得到结果:%IncMSE IncNodePurity x.glu 68.8034199 42207.351 x2.age 22.6784331 18569.370 x2.sex 6.2735713 2808.346 x2.bmi 1379.0675134 371372.494 x2.map 331.3925059 113411.547 x2.tc 18.6080948 14990.179 x2.ldl 24.3690847 17457.214 x2.hdl 216.2741620 64627.209 x2.tch 419.0451857 93688.855 x2.ltg 1514.0912885 379235.430 x2.glu 81.7568020 51984.121 x2.age.2 1.5242836 19364.582 x2.bmi.2 75.6345112 53635.024 x2.map.2 5.9156799 23049.475 x2.tc.2 1.6792683 15631.426 (省略部分输出) 其中第二列为均方误差递减意义下的重要性,第三列为精确度递减意义下的重要性。 六、其他的回归方法 除去我们所说的以上4种方法外,还有人工神经网络回归,SVM回归,他们可以通过nnet,rminer包中有关函数实现,这里我们从略。 在结束本文之前,我想我们可以做一件更有意义的事。在介绍diabetes数据集时,我们提到了这个数据最早是一个关于偏最小二乘的例子。那么想必他与这个模型的契合度还是不错的,我们可以尝试去算算利用偏最小二乘得到的训练集与测试集的NMSE。 代码如下:
- library(lars)
- library(pls)
- NMSE<-rep(0,5)
- NMSE0<-NMSE
- for(i
in 1:5){ - m<-index1[index2==i]
- reg.pls
<- plsr(y ~ x2, 64, data = diabetes[-m,], validation ="CV") - y0<-predict(reg.pls,diabetes[-m,])
- y1<-predict(reg.pls,diabetes[m,])
- NMSE0[i]<-mean((diabetes$y[-m]-y0)^2)/mean((diabetes$y[-m]-mean(diabetes$y[-m]))^2)
- NMSE[i]<-mean((diabetes$y[m]-y1)^2)/mean((diabetes$y[m]-mean(diabetes$y[m]))^2)
- }
运行结果:测试集NMSE为0.6094071,训练集NMSE为0.4031211。这里也仅仅是得到了一个优于回归树,与bagging回归相当,不如随机森林的一个结果。也说明了机器学习方法在实际问题中还是有一定优势的。但这绝不意味着这样的回归可以代替OLS,毕竟这些数据挖掘的方法在表达式的简洁程度,计算量等诸多方面还是存在着或多或少的不足。更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容







所有评论(0)